はじめに
こんにちは。
岩津です。
弊社では福利厚生の一環…というわけではないと思いますが、NvidiaのGPUワークステーションを社内から技術部門のみならず誰でも利用することができます(申請さえすれば)。
どういうものかというと、こういうものです。↓
過去ブログ参照:DGX-STATIONを導入しました!
環境が用意されているというのは非常に大事なところではあるものの”誰でも触れる”といっても現実的にはハードルが高いところもあったりするので、広く一般というのはなかなか難しいところですね。
そんな状況を改善すべく今回は前回の続きで『GPUワークステーションをもっと活用する』ことについて書きたいと思います。
今回の前提
- Singularityの導入ができている
- Jupyter Notebookが実行可能なSingularityコンテナイメージがある
- Docker環境はあるが利用ユーザーにスーパーユーザー権限は渡したくない
- Dockerのバージョンが18.09(事情により19系に上げられない)
対象とする読者
- GPUワークステーションを使ってなにかやりたい弊社社員
- Jupyter Notebookを使っているがコマンドライクに実行して帰りたい人
- 共用のNotebook環境に限界を感じている人
流れ
- 弊社Jupyter Notebookの推奨環境について
- Singularityを利用したJupyter Notebookの起動
- Notebookをコンテナごとコマンドで実行する
かなり読者ターゲットが絞られ環境依存が強いですね。
Singularityを利用されることを検討されている方には少しでも参考になる部分があればいいなと思っております。一つご容赦ください。
弊社Jupyter Notebook推奨環境について
まずは弊社社内で利用開放しているJupyter Notebookの環境について説明します。
社内の一般的な利用者にはまずこれを利用してもらう推奨環境となります。
ワークステーションではJupyterHubを利用し、利用者には各個人用コンテナ上のNotebookにブラウザでアクセスしてもらっています。
|
1 2 |
http://(ワークステーションのIPアドレス)/ |
にアクセス後、自分のIDとパスワードを入力すると自分の用のJupyter Notebookが利用できます。
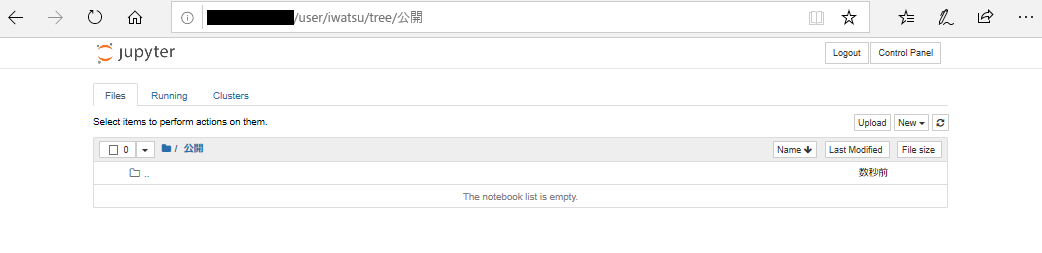
ワークステーション上の自分のホームディレクトリがマウントされた状態ですので、必要なデータやプログラムは自分のディスク領域に保存することができます。
- JupyterHub + Docker Engine
を利用しているので構成としては以下のようになりました。
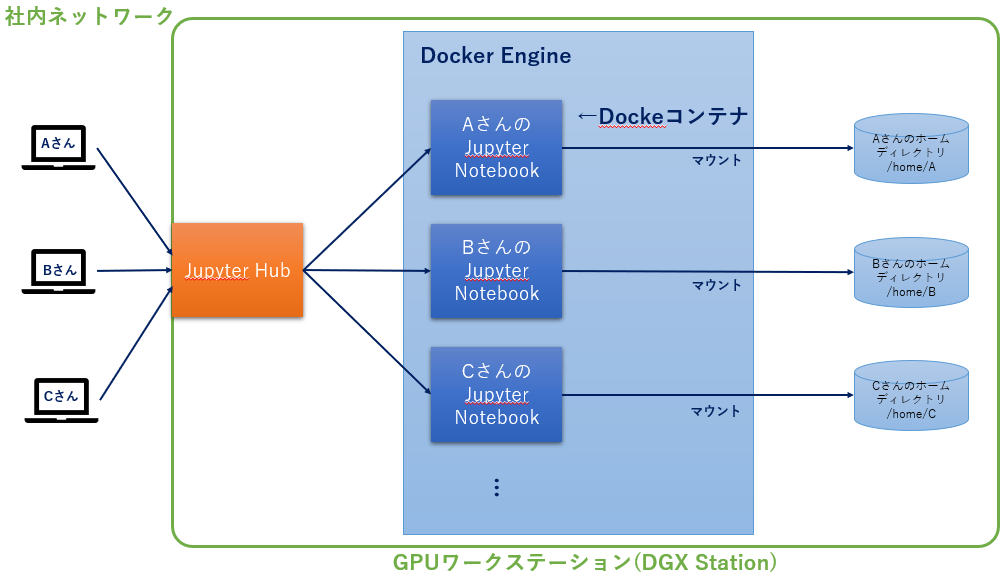
この構成の利点は
- 各ユーザーのデータやプログラムを共用ディスクに置かなくて良いので、プライバシーが守られる
- 人のファイルを壊してしまう恐れがない
- Jupyter Notebook環境が不調になってもユーザー単位でコンテナの再起動ができる
- 共通のコンテナイメージを利用すればメンテコストが低減できる
- JupyterHubがユーザーがログインしたときにコンテナを立ち上げてくれる
などがあります。
対して不満としては
- コンテナイメージは共有のものを利用しているため、新しいライブラリを含んだイメージを作成した場合、JupyterHubの再起動を必要とする運用となっている
- JupyterHubの再起動を行う場合、全コンテナを削除する運用となっている
というものがあります。
回避策はいくつかあると思うのですがコンテナイメージの管理を煩雑にしたくないのもあり今は新しいイメージができるたびにJupyterHub、Jupyter Notebookの環境を再起動しています。
当然利用者からは 「データ処理中なので再起動勘弁して」 などの声が出てくるので再起動するのはタイミングを見計らってとなってしまいます。
ちょっと嫌ですね。
Singularityを利用したJupyter Notebookの起動
そんな利用者には前回のやり方で、ローカルPCではなく今度はGPUワークステーション上でSingularityを利用してJupyter Notebookを起動してもらうようにしました。
構成は以下となりました。
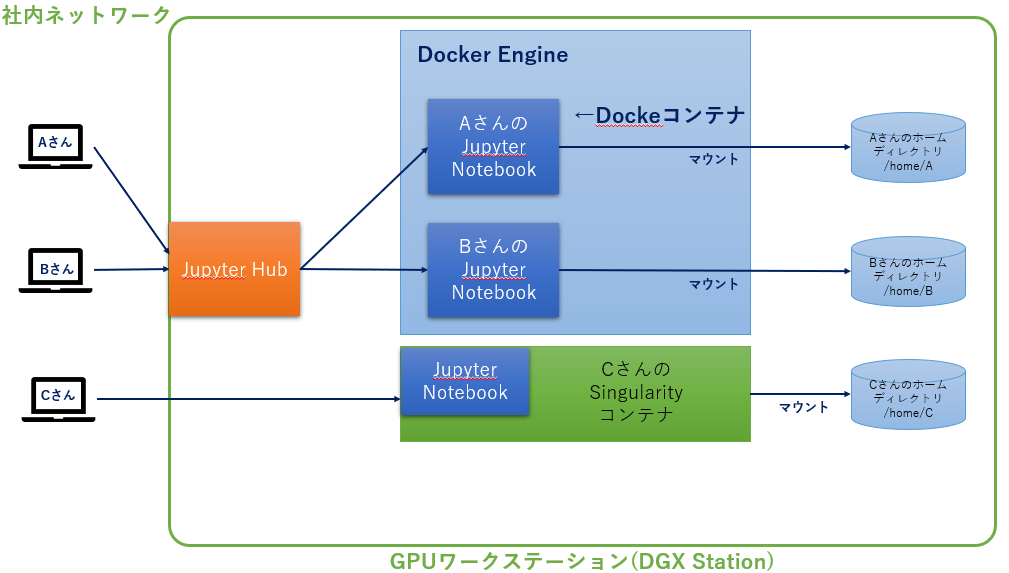
DockerイメージからSingularity用のコンテナイメージに変換したものを用意し、Singularityの起動は各ユーザーに以下のようにコマンドから起動してもらいます。
|
1 2 |
singularity exec --nv [コンテナイメージ] --port [ポート番号] |
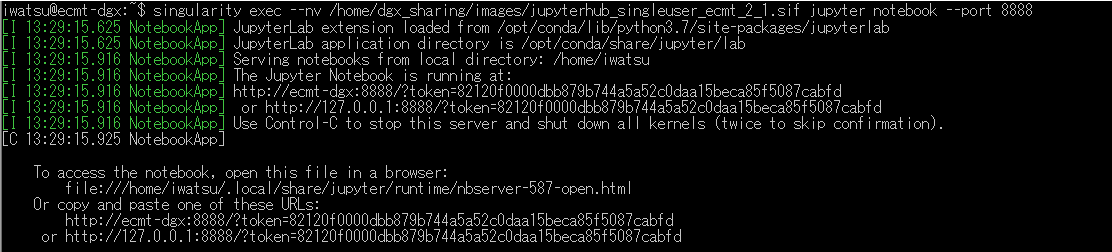
※コンテナイメージは各自必要なライブラリが入ったものを利用
少なくともこれで他の人のJupyter環境から切り離して実行できました。
これでCさんのタスク状況によらずJupyter環境の更新ができます。(逆もまた然り)
Notebookをコンテナごとコマンドで実行する
Jupyter Notebookを独立した環境で稼働させることができると、そこで様々な試行錯誤がやりすくなります。
ですが、データに対してどのような処理をするべきか、利用するモデルはどうするかなどひとしきりトライして方針がある程度きまったとしても、異なるデータを扱ったときに期待する結果が得られるかはまた別の話だったりします。
となると次に欲しくなってくるのは以下のような要求です。
- データの条件(対象・範囲など)を手軽に変えて何度でも実行できるようにしたい
- 条件を変えた処理を複数並行で実施したい
- できれば環境としてはそれぞれ独立させておきたい
今回は上記を満たすため『コマンドからSingularityを起動する際のパラメータに処理対象のファイル(ipynbファイル)を指定し、コンテナ中のNotebookでコマンド処理させる』という手法をとりました。
私)「いっそ条件ごとにSingularityコンテナ立ち上げてJupyter Notebookをコマンド実行しちゃいましょう」
以下のような流れとなります。
簡単なNotebookを作成
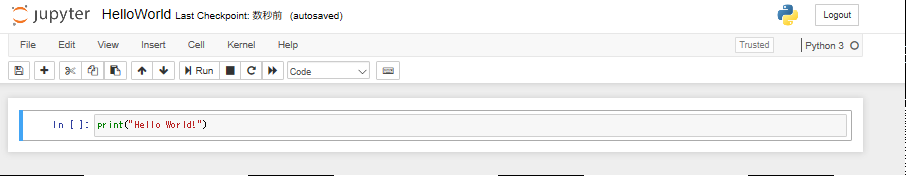
※HelloWorld.ipynbとして保存
Singularity起動時に”jupyter nbconvert”を実行
Notebookのコマンド実行にはnbconvertを利用し、Singularityに以下のように渡しました。
実行するHelloWorld.ipynbはSingularityがマウントする自分のホームディレクトリ配下に置いています。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 |
$ singularity exec --nv [コンテナイメージ] jupyter nbconvert --to asciidoc --ExecutePreprocessor.timeout=-1 --stdout --execute /home/iwatsu/sandbox/HelloWorld.ipynb [NbConvertApp] Converting notebook /home/iwatsu/sandbox/HelloWorld.ipynb to asciidoc [NbConvertApp] Executing notebook with kernel: python +*In[1]:*+ [source, ipython3] ---- print("Hello World!") ---- +*Out[1]:*+ ---- Hello World! ---- |
“Hello World!”が出力されているのを確認。
画像を出力してみる
次に実行結果として画像などを生成するケースをやってみます。
弊社の利用しているイメージにはmatplotlib basemapが予めインストールされているので、地図画像を表示してみました。
参考元
https://matplotlib.org/basemap/users/geography.html
実際のコード
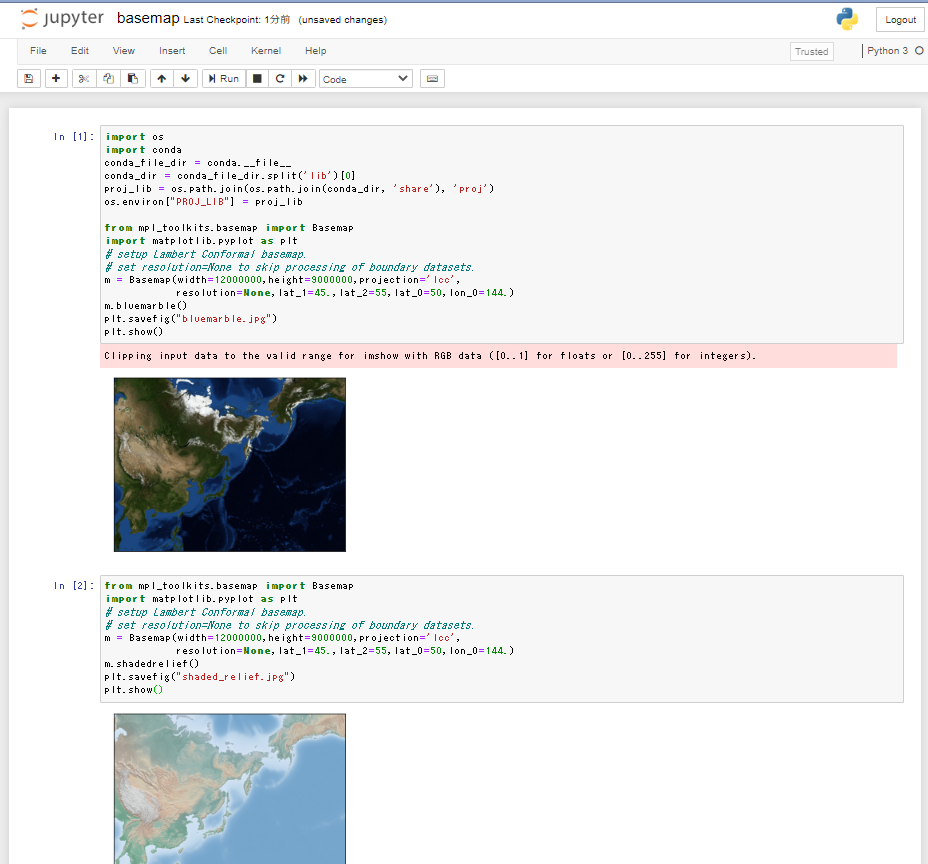
実行結果
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 82 83 84 85 |
iwatsu@ecmt-dgx:~$ singularity exec --nv /home/dgx_sharing/images/jupyterhub_singleuser_ecmt_2_1.sif jupyter nbconvert --to asciidoc --ExecutePreprocessor.timeout=-1 --stdout --execute /home/iwatsu/sandbox/basemap.ipynb [NbConvertApp] Converting notebook /home/iwatsu/sandbox/basemap.ipynb to asciidoc [NbConvertApp] Executing notebook with kernel: python3 +*In[1]:*+ [source, ipython3] ---- import os import conda conda_file_dir = conda.__file__ conda_dir = conda_file_dir.split('lib')[0] proj_lib = os.path.join(os.path.join(conda_dir, 'share'), 'proj') os.environ["PROJ_LIB"] = proj_lib from mpl_toolkits.basemap import Basemap import matplotlib.pyplot as plt # setup Lambert Conformal basemap. # set resolution=None to skip processing of boundary datasets. m = Basemap(width=12000000,height=9000000,projection='lcc', resolution=None,lat_1=45.,lat_2=55,lat_0=50,lon_0=144.) m.bluemarble() plt.savefig("bluemarble.jpg") plt.show() ---- +*Out[1]:*+ ---- Clipping input data to the valid range for imshow with RGB data ([0..1] for floats or [0..255] for integers).  ---- +*In[2]:*+ [source, ipython3] ---- from mpl_toolkits.basemap import Basemap import matplotlib.pyplot as plt # setup Lambert Conformal basemap. # set resolution=None to skip processing of boundary datasets. m = Basemap(width=12000000,height=9000000,projection='lcc', resolution=None,lat_1=45.,lat_2=55,lat_0=50,lon_0=144.) m.shadedrelief() plt.savefig("shaded_relief.jpg") plt.show() ---- +*Out[2]:*+ ----  ---- +*In[3]:*+ [source, ipython3] ---- from mpl_toolkits.basemap import Basemap import matplotlib.pyplot as plt # setup Lambert Conformal basemap. # set resolution=None to skip processing of boundary datasets. m = Basemap(width=12000000,height=9000000,projection='lcc', resolution=None,lat_1=45.,lat_2=55,lat_0=50,lon_0=144.) m.etopo() plt.savefig("etopo_relief.jpg") plt.show() ---- +*Out[3]:*+ ---- Clipping input data to the valid range for imshow with RGB data ([0..1] for floats or [0..255] for integers).  ---- +*In[ ]:*+ [source, ipython3] ---- ---- iwatsu@ecmt-dgx:~$ |
生成された画像
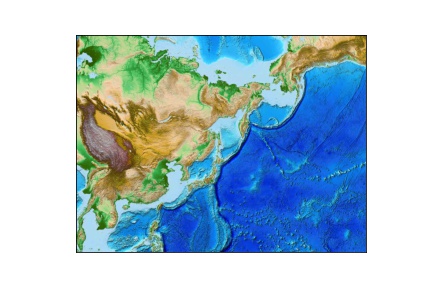
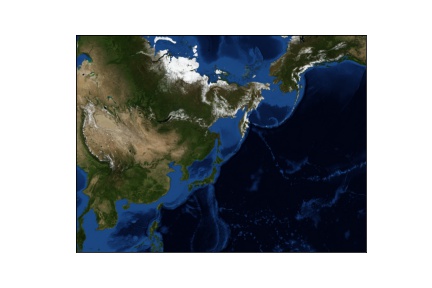
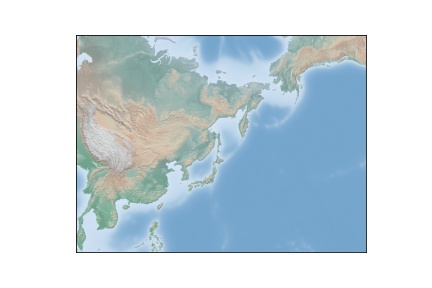
生成された画像が確認できました。
まとめ
- Jupyter NotebookをDockerで運用しているなら、Singularityでも移行できる
- Singularityを利用してNotebookをコマンドライクに実行できた
- コマンド実行できるので条件の異なる処理を手軽に実行できる
- 【社内連絡】推奨環境で慣れたらSingularity使って独自コンテナイメージの利用できます。どんどん使ってください。